提起虎(Panthera tigris),我们脑海中都会浮现出一个经典的大猫形象——巨大的身形,壮硕的头颅,肌肉虬结的四肢,橙黄毛皮上独特的黑色条纹。无论在历史典籍中还是影像资料里,野生的虎似乎都有着相似的形象。那么,我们常说的东北虎、华南虎、孟加拉虎等等,究竟有啥区别呢?这些称谓不同的老虎之间,又有着怎样的身世沉浮?

虎的经典形象。图片来源:Brocken Inaglory, Wikimedia Commons
答案就在它们的基因组中。近日,北京大学基因组多样性与演化研究组的团队在《现代生物学》上发表了一篇新的研究论文。他们利用全基因组测序技术,对32只虎的遗传信息进行了分析,揭示了虎这个物种在各个地区的演化历史和亚种分类依据[1]。
分布广泛的大猫
作为巨型的猫科动物,虎的分布范围其实非常广泛:西到南亚次大陆的印度和孟加拉国,南至巽他群岛中的苏门答腊岛,向东向北远达中俄交界的远东地区,都是这些百兽之王的疆域。虽然野生虎由于栖息地与人口密集地区的重合而备受生存威胁,但它们至今仍在这些地区保有着碎片化的栖息地。
历史上,虎的身影还曾出现在东南亚的爪哇、巴厘岛,甚至中亚的里海沿岸,这意味着这个物种适应了热带雨林、季风雨林、亚热带/温带森林、针叶林甚至是中亚干旱地区的绿洲等各种生境。
人们很早就对不同地区的虎进行了区分。到1968年,现存的6个虎亚种当中的5个已经得到了命名,包括指名亚种(就是亚种名和种名相同的)孟加拉虎(Panthera tigris tigris),分布在中南半岛的印支虎(P. t. corbetti),以及苏门答腊虎(P. t. sumatrae),华南虎(P. t. amoyensis)和东北虎(P. t. altaica)。在2004年,根据基因组中的部分多样性位点信息,研究者发现位于马来半岛地区的虎个体与其它印支虎有所分化,并以此命名了新的虎亚种马来虎(P. t. jacksoni) [2]。

马来虎亚种。图片来源: B_cool,Wikimedia Commons
亚种划分的困难
在生物学研究中,“亚种”的概念并非明确易行。从分类学上来说,亚种是比物种更低一级的分类阶元,著名遗传学家Stephen O’Brien与进化生物学家Ernst Mayr于1991年共同提出了关于生物亚种定义的原则,将其定义为“一定地理区域内分布的与同种的其他亚单元在分类学上有差异的种群的组合” [3]。
与物种概念不同,不同亚种的动物个体并不存在生殖上的隔离,相邻亚种种群存在自然状态下的交汇也不足为奇,然而亚种之间的区别通常包括一定的地理隔阂和由此导致的遗传信息、形态学特征和自然历史的分化。有意义的亚种定义,需要能够体现出真正的“分隔”——比方说,分属不同亚种的两只老虎,应该要比同一个亚种内部的两只老虎有着更多的遗传差别。
在没有分子生物学手段的时代,研究者们只能通过遗传信息的宏观体现——生物体的各种形态学特征,来进行亚种的定义和分类。对于虎这个物种来说,历史上定义的这些亚种之间并没有显著的形态学差异。因此,在2015年,有研究提出应该把亚洲大陆上的所有虎亚种合并,而原本存在于巽他群岛上的虎也应该统一[4] 。这样一来,虎这个物种的现存亚种数量就会从六个变成两个——“大陆虎”和“巽他虎”。

摄于1899年的里海虎影像资料。按照两个亚种的分类规则,这应该是一只“大陆虎”。图片来源:Wikimedia Commons
然而,形态学上的相似并不能证明这些虎演化历史上的雷同,因为遗传差异并不总会反映在外表形态上。在最新的研究中,罗述金团队及其合作者采用了已知地理来源的32只野生虎作为研究样本,对这些个体进行了全基因组的测序,希望直接从遗传信息的尺度检验历史上定义的六个亚种间是否存在显著的分化。
基因组中的分家故事
用分子数据推测演化历史,靠的是比对多个基因组中的相对差异。基因组本质上是上亿个核苷酸小分子相连组成的大分子序列,其中ATCG四种核苷酸的不同排列组合就蕴含着遗传信息。在漫长的演化过程中,各种随机的遗传变异不停地发生,表现为基因组中的一些核苷酸发生了增减替换。因此,如果两只老虎的基因组序列差别较小,这意味着它们的分化较小,亲缘关系更近,反之则表明二者的共同祖先更加久远,或者说二者所代表的虎的群体“分家”历史比较长了。
基于这个最基本的原理,研究者根据一系列算法分析指出,32只来自不同地区的虎可以分成六组,分别对应之前提到的六个亚种。比如说,在不同个体中,单个核苷酸位点上出现两种不同的核苷酸是一种遗传差异(单核苷酸差异,SNV)。在虎的线粒体基因组中,仅有196个SNV位点,而其中有173个的差异和亚种划分相对应——不同的亚种有着不同的核苷酸,而相同亚种的个体核苷酸相同。这说明六个亚种之间的地理隔离的确造成了遗传上的显著“分隔”,也就是说亚种间的区分是有意义的。
利用基因组序列信息,研究者们还对虎的种群历史进行了统计学推断,揭示了这个物种与地质变化息息相关的演化历史——从两三百万年前,虎就作为一个独立物种与豹属其它的大猫分道扬镳,开始了独自分化。然而,统计推断表明现有的这32个基因组可以追溯到11万年前的共同祖先。换句话说,从虎成为独立物种开始到11万年前,这段演化历史中产生的绝大多数遗传变异都没有保留下来。这是为什么呢?
种群遗传学中有个所谓的“瓶颈效应”,说的是当动物群体数量大幅减少时,原本存在于群体中的遗传差异也随之丢失。那么,这些虎在当时经历了什么呢?原来,11万年前,地球刚刚经历了长时间冰期肆虐的严寒天气,适应森林生境的老虎们很可能正是遭遇这个“瓶颈”,导致种群数量减少,遗传多样性也随之降低。
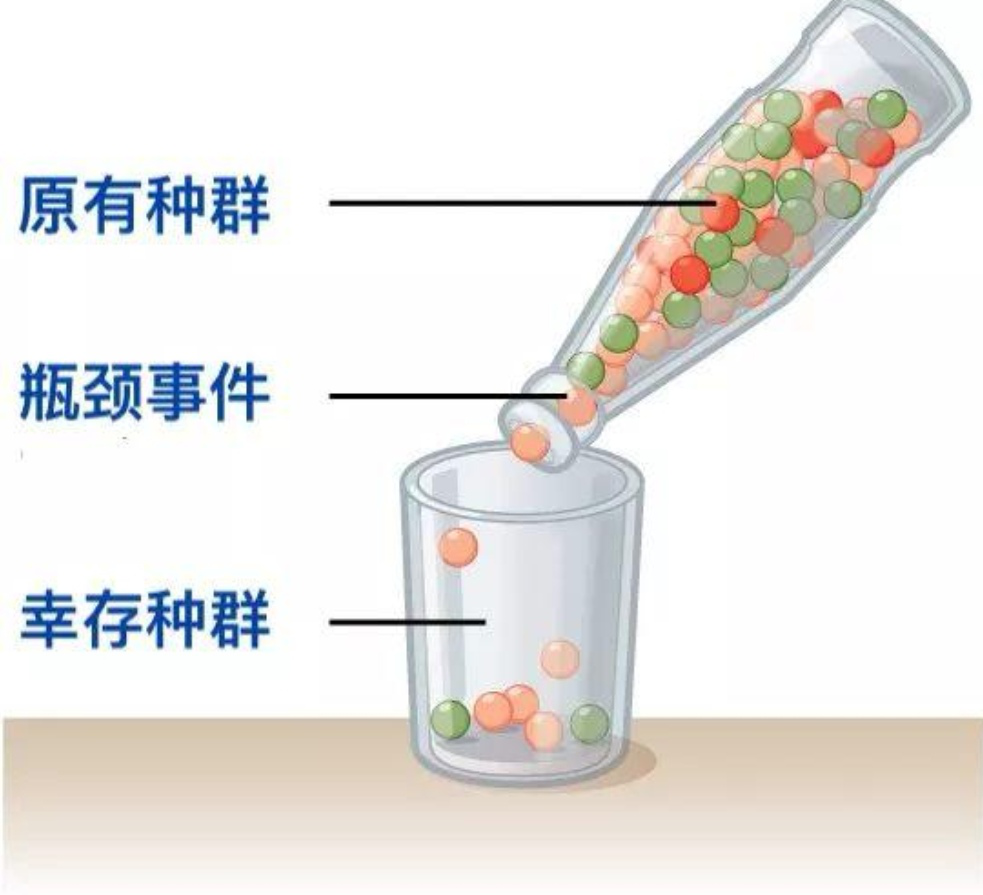
瓶颈效应示意图,经过瓶颈筛选后,红色的个体彻底消失了。图片来源:Wikimedia Commons汉化:卢平
寒冷气候还有另一个效果:在温暖时期遍布亚洲的森林,很可能逐步退缩到了一些互不相连的地区碎片中,也就是所谓的“避难所”。“逐森林而居”的虎也自然因此被分割成了不同的群体分别演化,这也许就是11万年前各个亚种分化的起始。统计推断出的演化历史,也展现了虎在末次冰期的气候动荡中,逐步占领各个地区的筚路蓝缕。
在东南亚的生物演化史上,苏门答腊岛上的多峇超级火山是个狠角色。大约75000年前的喷发导致了全球性的气候变冷,也很可能导致巽他群岛上原有的老虎团灭。现存的苏门答腊虎和曾经的爪哇虎、巴厘虎,正是在距今67000年前后由印支地区(中南半岛和马来半岛)迁入岛屿重新“殖民”的个体。
此后,印支地区的虎又在距今53000年前后进入南亚,分化出了孟加拉虎。大约34000年前,沿西南地区进入中国的老虎一面向北扩散形成后来的东北虎,一面在西部遇到了此前蜗居在我国西南山地的一小支遗老遗少,形成了华南虎亚种。而随后的末次冰期最高峰,使得克拉地峡的森林退化,对印支地区的个体形成阻隔,分化出了印支虎和马来虎。大概12000年前,雄踞北方的东北虎向西探索,形成了遗传上与其差异不大的里海虎亚种。
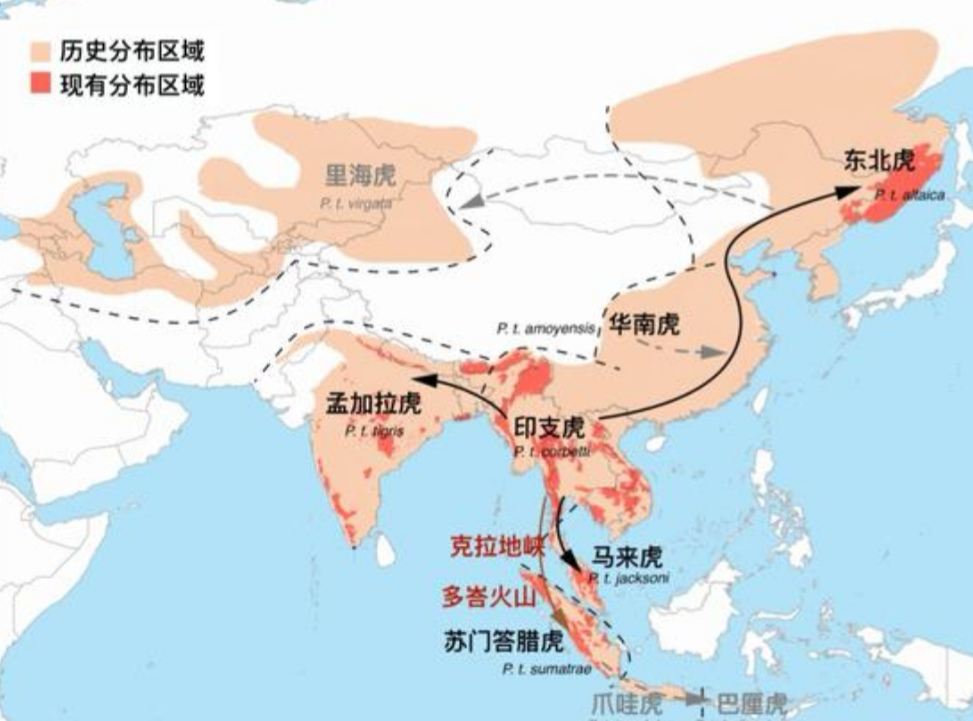
虎的历史分布及现有分布区域,箭头表示论文中推测的各亚种演化历史。供图:北京大学生命科学学院 刘悦晨,汉化:卢平
亚种演化的意义
虽然难分彼此,但对比苏门答腊虎和亚洲大陆上的各个亚种可以发现,前者平均体型略小而毛色略深。除了种群历史,研究组也检测了基因组中经历过自然选择的区域, 与体重相关的ADH7、与色素合成相关的UQCRQ以及与胰岛素代谢相关的EXOC5等基因在二者之间有着显著增多的遗传差别,这表明这些基因在演化过程中受到了选择作用而保持了特定的功能。考虑到岛屿雨林生境中光照更少、缺乏大型有蹄类猎物,小身板和更隐蔽的深色毛皮也许是苏门答腊虎对当地环境的一种适应。
那么,了解老虎们分家的故事有什么意义呢?说起来,野生动物保护的最主要目标就是保护“遗传多样性”,而当我们知道远东地区的虎和南亚地区的虎其实“不一样”,才能制定与之相应的保护策略,对来自不同地区的虎分别管理。一个例子是,亚种的区分可以让我们避免原本有所不同的群体在动物园的繁殖计划中过度混合 ,丧失各自的遗传特色。
更重要的是,正如我们在苏门答腊虎的案例中看到的那样,不同地理分布的亚种都在不断地适应其特定的生态环境,随着时间的推移,积累的适应性特征逐渐增多,与其它亚种的分化也就逐渐增大。
正如达尔文在1886年的《物种起源》中所说,每一个亚种都具有最终演变成为物种的可能。尽管我们无法预知哪一个特定亚种将具有这样的可能性。但是我们有充分的理由对亚种进行保护,因为这就是在保护自然的演化过程本身和演化的成果。挖掘这些基因组背后的身世之谜,也正可以告诫我们,不能对任何虎亚种的衰退掉以轻心——因为,它们各有各的故事,各有各的精彩。
参考文献:
Liu et al., Genome-Wide Evolutionary Analysis of Natural History and Adaptation in the World’s Tigers, Current Biology (2018), https://doi.org/10.1016/j.cub.2018.09.019
Luo et al. Phylogeography and genetic ancestry of tigers (Panthera tigris). PLoS biology (2004), 2(12), e442.
O’Brien SJ, Mayr E. Bureaucratic mischief: recognizing endangered species and subspecies. Science (1991), 251, 1187–1188.
Wilting et al. Planning tiger recovery: Understanding intraspecific variation for effective conservation. Science Advances (2015). 1, e1400175.
